Den här artikelserien,
som började vid midsommar och slutar nu, har tittat in på flera
raser och deras olika genetiska problem. Lundehund. Doberman. Boxer. Bullterrier. Collie.
Här möter ni flera av
dem igen - plus ett antal andra - i ett blogginlägg, som liknar en svensk deckare. Man trampar sig mödosamt fram genom alla beskrivningarna i inledningen, men de gastkramande grejorna kommer på slutet. Det handlar om verkliga inavelsgrader. De är inte vad vi tror att de är.
Förr fanns ett enda sätt
att beräkna inavelsgrad eller COI, coefficient of inbreeding, som är
den andel av ett djurs gener som i genomsnitt är identiska med
generna hos andra djur av samma ras med sluten stambok, alltså mera
eller mindre släkt. Man fick använda stamtavlan. Så görs det
fortfarande i angivandet av COI – standard är COI beräknad på
tre, eller fem, eller sex, generationers stamtavla; någon enstaka
gång på tio. Att gå längre bak än så kräver mycket tid
och/eller datorkapacitet. Samvetsgranna uppfödare har legat på mage
på golvet med enorma pappersark och många kvällars jobb framför
sig och ändå inte haft en rimlig chans att få till det över mera
än några generationer. För även om man har tiden och tålamodet,
blir namnen och generationerna och släktskapsbanden mellan dem för
många. Och framförallt bygger metoden på att namnen på papperet
finns där, att förfäderna går att hitta, hela vägen bakåt till
rasens första grundare, och att det är rätt namn - och det finns
ingen garanti för någondera. Om det finns en tom plats i
förfädersleden elva generationer längre bak på ena sidan och på andra sidan fjorton
generationer längre bak, vad skall den stackars uppfödaren göra
åt saken? Och vad händer om den där rätt så obesläktade hanen
från en annan kennel fem led bakåt i stamtavlan i själva verket
var tikens bror eller far? Oavsett om det berodde på en uppfödare,
som ville ”prägla” avkomman lite extra (vilket förmodligen har
hänt ) eller om det berodde på hundars utmärkta förmåga att fixa
sina parningar på egen hand (vilket alldeles säkert har
inträffat), så blir inavelsberäkningen fel.
Det är säkrare att mäta
direkt från verkligheten och titta på de hundar som lever idag. Man tittar på DNA. DNA
kan inte ljuga, som en genetiker sa.
Sedan några år tillbaka
kan man använda två metoder till förutom stamtavlan. Båda är
baserade på DNA och har en sak gemensam: de ger alltid högre värden
för släktskap än vad stamtavleberäkningarna gör.
Den ena metoden använder
sig av något som kallas SNP, single nucleotide pairs, och jämför
vissa enskilda baspar i ett antal gener hos flera hundar av samma ras. Är
de ofta olika? Skiljer de sig lite ibland? Är de ofta identiska? Man
försöker undvika gener i områden, som man redan vet är likadana
hos alla hundar av rasen – om man till exempel skulle få för sig
att titta på SNP i området för genen för svart pälsfärg i en
ras där varenda hund är svart, skulle man få ett orättvist högt
värde för likadana SNP och tro, att hundarnas genetiska
likformighet var större än den är. De tidiga beräkningarna av SNP
använde jämförelsevis få baspar och anses idag ge för grova
mått: man fångar inte så mycket fisk med nät, som har väldigt
stora maskor. De senaste använder fler baspar och ses som mera träffsäkra.
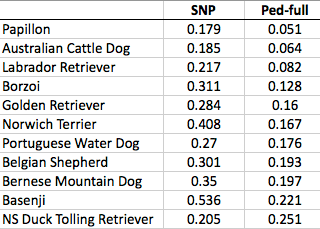
Tabellen visar exempel på skillnader i inavelsgrad för några olika
raser beroende på om man beräknar utifrån stamtavlan i alla
generationer eller mäter direkt från DNA med SNP-metoden över 173 600 olika baspar.
|
Tabellen är hämtad från Carol Beuchats inlägg Inbreeding of purebred dogs determined from DNA och publiceras med hennes tillåtelse. Ped-full = stamtavleCOI beräknad på alla generationer från rasens början
Ni ser själva, att
skillnaderna är stora mellan stamtavleanalysen och de
faktiska hundarnas DNA. Enda rasen som har högre inavelskoefficient
på papperet än i DNA-mätningarna är den allra yngsta rasen, den
mest kortast sluten stambokstid och lägst antal generationer. ACD har i helstamtavleanalysen en inavelskoefficient på 6,4%, men DNA mätt via SNP visar på 18,5%. Golden, i USA precis som här en av
de mest populära raserna, har hos de genomgångna hundarna en
stamtavlebaserad inavelsgrad på 16%. Det kan man tycka är illa nog,
eftersom det ligger över halvsyskonparning i släktskap; men
SNP-analysen hittar genetisk samstämmighet i drygt 28%. Basenji ser
ut att ha en inavelsgrad på drygt 22% räknat på hela stamtavlan
tillbaka till ett fåtal importerade afrikanska ursprungshundar, men
53% beräknat från SNP! Det finns som sagt inga ord i vårt språk
för en sådan grad av släktskap. Men någonstans läser jag, att en
COI på 50% motsvarar tre generationers helsyskonparningar.
* * *
Beuchats tabell ovan baseras på en en
vetenskaplig rapport publicerad i tidskriften Company of
Biologists i slutet av 2016:
RESEARCH ARTICLE
Whole genome sequence, SNP
chips and pedigree structure: building demographic profiles in
domestic dog breeds to optimize genetic trait mapping
Dayna L. Dreger, Maud Rimbault, Brian
W. Davis, Adrienne Bhatnagar, Heidi G. Parker, Elaine A. Ostrander
Disease Models & Mechanisms 2016 :
dmm.027037 doi: 10.1242/dmm.027037 Published 17 November 2016
Inavelsgraden för 80
renraser bedömdes alltså på tre sätt. Först gjordes en
stamtavleanalys på fem generationer, tio generationer och slutligen
hela stamtavlan av tio hundar i varje ras. Sedan gjorde man en
uppskattning av genetisk likformighet, homozygoti, med SNP-metoden på
samma hundar i varje ras. Slutligen gjorde man en hel genomanalys, en
WGS (whole genome sequencing) av en hund från varje ras–
sammanlagt 800 hundar från 80 AKC-raser.
Och så jämförde
man resultaten med varandra. Det är poängen med den
här studien: man samkör tre olika metoder. En metod
som man vet har lägre säkerhet, stamtavlan; en metod med högre men
fortfarande otillräcklig säkerhet, SNP-analysen; och en metod som
inte kan visa fel, men som
man (av kostnadsskäl) bara gör på en hund, alltså avläsning av
hela den genetiska koden med kontroll av hur långa sträckor av
homozygota (dubblerade) gener som finns där. Långa sträckor av
homozygoti (förkortat RoH, runs of homozygosity) skapas genom
inavel. Ju intensivare och ju längre man har inavlat, desto längre
blir de likformiga genavsnitten.
När man ställer upp de tre metoderna intill varandra, vad blir det då för resultat? Rafflande.
Bodil Carlsson
Inga kommentarer:
Skicka en kommentar